Heute erklären wir Ihnen, wie sich eine Varianzanalyse (ANOVA) in R umsetzen lässt.
Die Daten
Bei dieser Auswertung verwenden wir den Datensatz iris. Dieser wird einer einer R-Session direkt in den Workspace geladen. Sie können als unmittelbar auf diesen Datensatz zugreifen. Das Objekt hat auch den Namen iris
Der Datensatz umfasst Daten zu Blattflächen, -längen etc. bei 3 Irisarten. Wir möchten die KelchBbattlänge in Abhängigkeit der Irisart erklären. Da die abhängige Varible metrisch ist und die unabhängige katgeorial bietet sich die ANOVA zur Untersuchung auf Unterschiede in den Kelchblattlängen zwischen den Arten an.
Die Analyse
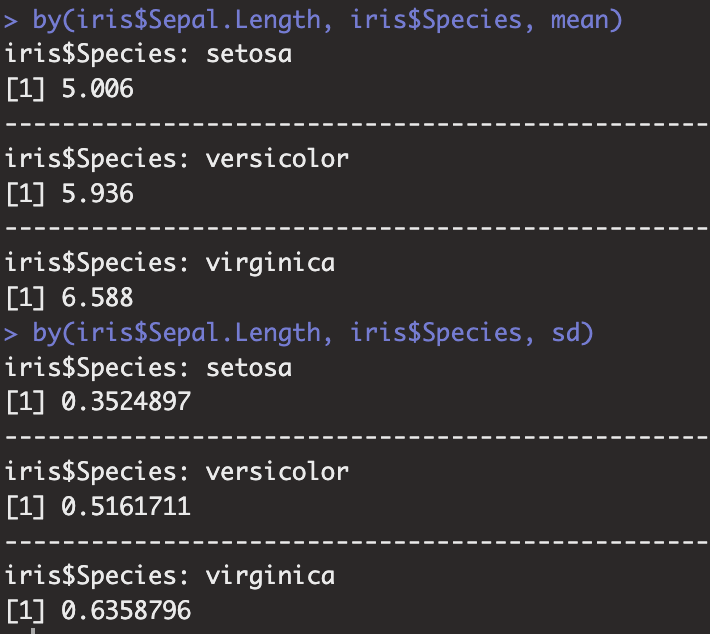
Zunächst betrachten wir die Mittelwerte zwischen den Sorten mittels deskriptiven Statistiken. Hierzu können wir die Funktion by nutzen by(iris$Sepal.Length, iris$Species, mean) und erhalten damit die Mittelwert in den Kelchblattlängen pro Spezies. Analog erhalten wir mit by(iris$Sepal.Length, iris$Species, sd) die Standardabweichungen. Kommendes Bild zeigt uns die Ergebnisse. Setosa hat hierbei den geringsten Mittelwert, M = 5,0, SD = 0.4. Hierauf folgt Versicolor, M = 5,9, SD = 0,5. Die Virginica hat mit M = 6,6, SD = 0,6 im Durchschnitt die längsten Kelchblattlängen.
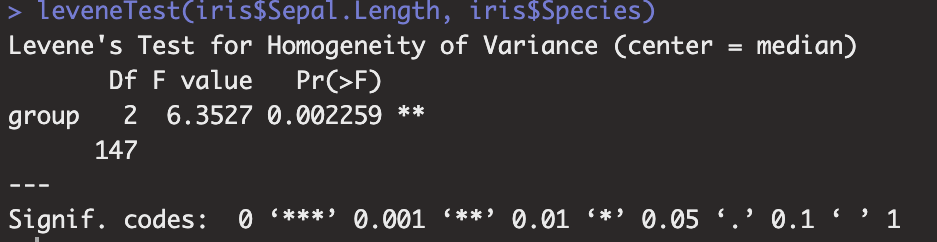
Die Varianzanalyse hat zwei zentrale Annahmen. Zum einen Varianzhomogenität und zum Anderen Normalverteilung der Residuen. Zunächst prüfen wir die Varianzhomogenität mittels des Levene-Tests. Im Paket car ist eine Funktion zur Berechnung des Levene-Tests implementiert. Laden Sie hierzu das Paket mit dem Befehl library(car). Die Funktion, welche wir nutzen rufen wir wie folgt auf: leveneTest(iris$Sepal.Length, iris$Species). Kommendes Bild zeigt die Ergebnisse. Es zeigt sich, dass die Varianzen signifikant unterschiedlich sind, F(2, 147) = 6,35, p = 0,002. Da unterschiedliche Varianzen vorliegen, ist eine Welch-Korrektur bei der ANOVA sinnvoll. Eine Funktion, welche die ANOVA mit Welch-Korrektur durchführt, ist oneway.test().
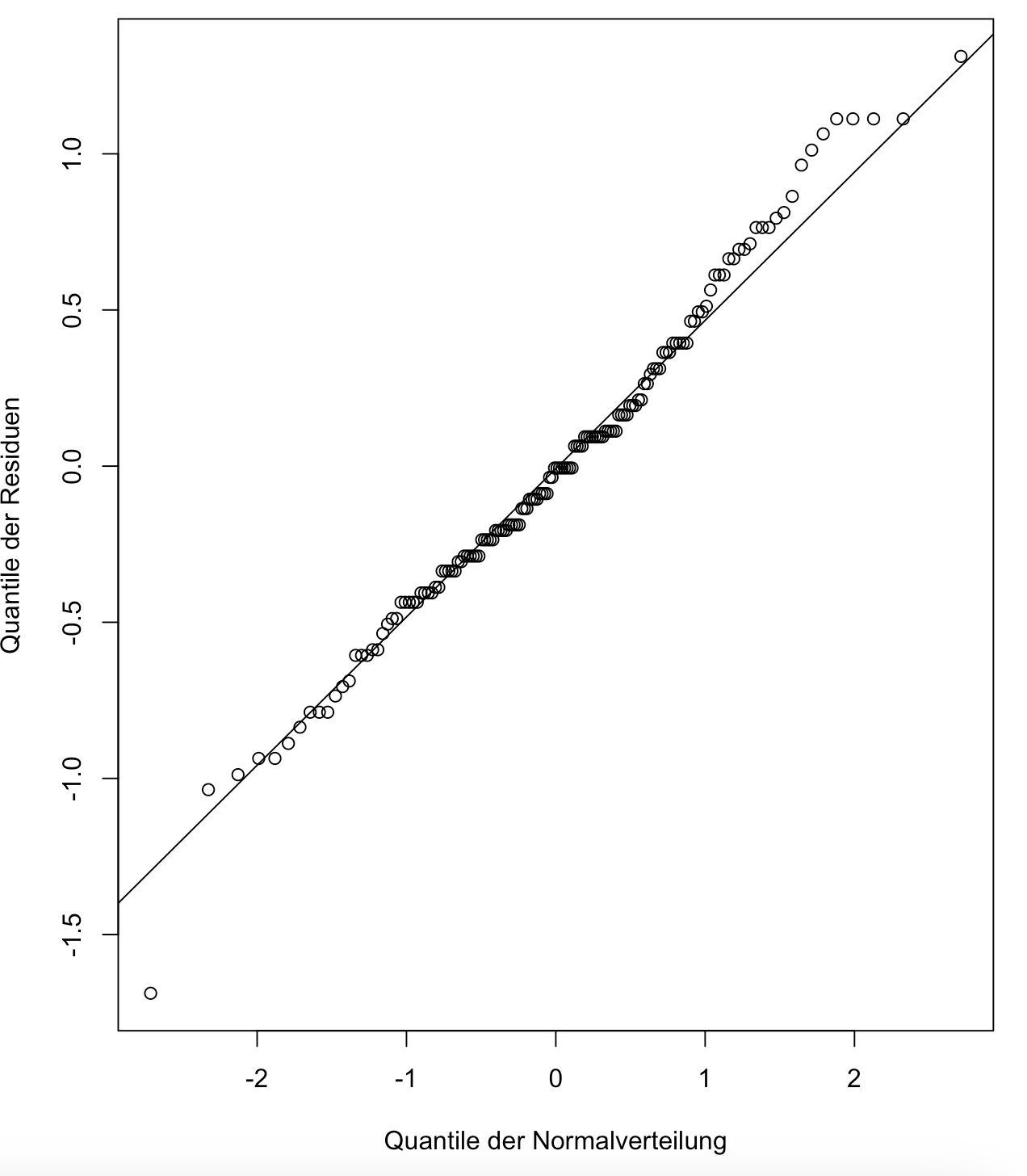
Zuvor untersuchen wir jedoch die Normalverteilungsannahme. Hierzu müssen wir zunächst die gewöhnliche ANOVA berechnen (ohne Welch-Test) und daraufhin die Residuen extrahieren. Mit model <- lm(Sepal.Length ~ Species, data = iris) spezifizieren wir unsere Untersuchungen als Regressionsmodell. Nun ist es möglich mit residuals(model) an die Residuen zu kommen und diese auf Normalverteilung zu untersuchen. Mittels qqnorm(residuals(model), ylab = "Quantile der Residuen", xlab = "Quantile der Normalverteilung", main = NULL) und qqline(residuals(model)) erhalten wir die kommende Grafik. Die Punkte konzentrieren sich dabei sehr nah an einer Linie, sodass Normalverteilung angenommen werden kann.
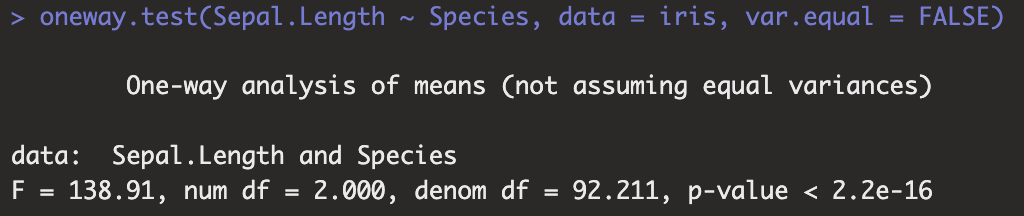
Die Annahmen der ANOVA wurden überprüft. Normalverteilung liegt vor, Varianzhomogenität jedoch nicht. Somit wird nun die ANOVA mittels eines Welch-Tests durchgeführt. Mit oneway.test(Sepal.Length ~ Species, data = iris, var.equal = FALSE) kann dann die Varianzanalyse mit Welch-Korrektur durchgeführt werden. Kommendes Bild zeigt das Ergebnis. Es zeigen sich signifikante Unterschiede in der Kelchblattlänge zwischen den Arten, F(2, 92,2) = 138,9, p = 0,0000.
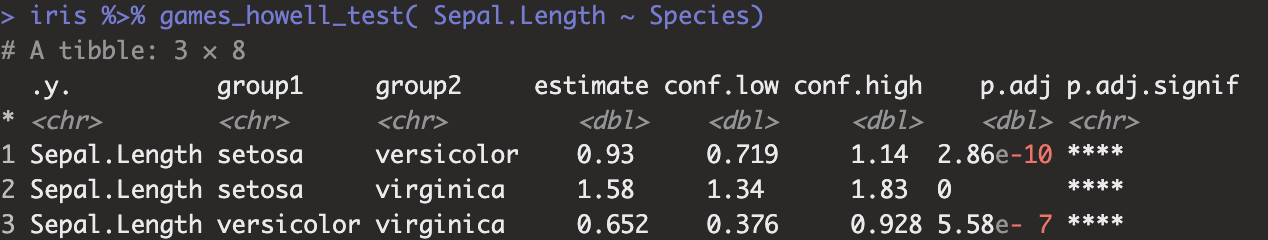
Da signifikante Unterschiede zwischen den Sorten bezüglich ihrer Kelchblattlängen vorlagen, wird nun in einer Post Hoc Analyse untersucht, welche Sorten sich paarweise unterscheiden. Auf Grund der verletzten Varianzhomogenität wird der t-Test nach Games-Howell gemacht. Im Paket rstatix ist eine Funktion implementiert. Laden Sie hierzu zunächst das Paket mit library(rstatix). Mit der Befehlsfolge iris %>% games_howell_test( Sepal.Length ~ Species) erhalten Sie dann den Post Hoc Test nach Games-Howell. Kommendes Bild zeigt die Ergebnisse. Es unterscheiden sich hierbei alle Sorten paarweise signifikant.
Sie interessieren sich für Analysen in R. Dann besuchen Sie unsere R-Hilfe.
